- Об обобщенных смешанных линейных моделях (GLMM) и о функциях R, которые могут их рассчитать
Вы сможете
- Построить обобщенную смешанную модель для бинарных данных.
- Применить для построения модели функции из нескольких пакетов, реализованных в R.
\(f(y; N, \pi) = \frac{N!}{y! \times (N-y)!} \times \pi^y \times (1 - \pi)^{N-y}\)
Среднее: \(E(Y) = N \times \pi\)
Дисперсия: \(var(Y) = N \times \pi \times (1-\pi)\)
Параметр \(N\) определяет количество объектов в испытании
Парметр \(\pi\) - вероятность события (\(y = 1\))
\(0 \le Y \le +\infty\), \(Y\) целочисленные
Дискретный отклик: 1 или 0
Вероятност события: \(\pi = \frac{N_i}{N_{total}}\)
Шансы (odds): \(odds=\frac{\pi}{1-\pi}\)
Логиты (logit): \(ln(odds)=\ln(\frac{\pi}{1-\pi})\)
Каноническая связывающая функция - логит-функция: \(\eta(\pi)=\ln(\frac{\pi}{1-\pi})\)
family = binomial(link = "logit")
Помимо логит-функции можно применить еще несколько:
Complementary Log-Log связывающая функция : \(\eta(\pi)=\ln(-\ln(1-\pi))\)family = binomial(link = "cloglog")
Пробит - связывающая фунция: \(\eta(\pi)=\Phi^{-1}(\pi)\)family = binomial(link = "probit")
Данные взяты из работы
Yakovis Y., Artemieva A. "Bored to Death: Community-Wide Effect of Predation on a Foundation Species in a Low-Disturbance Arctic Subtidal System" PLOS, 2015. DOI: 10.1371/journal.pone.0132973
Мы будем оценивать связь вероятности гибели балянуса от нападения Boreotrophon clathratus.
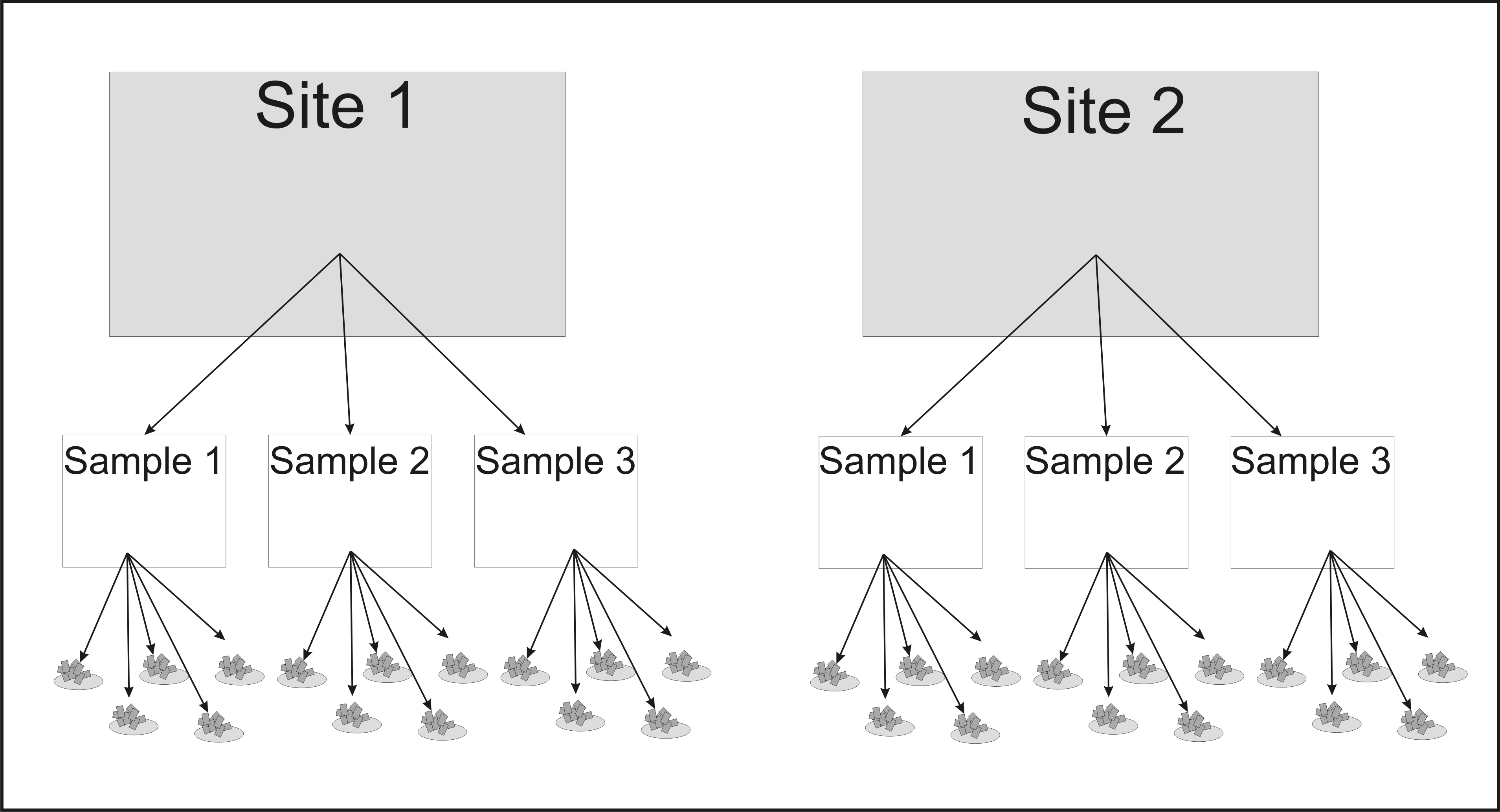
bal <- read.table("data/Yakovis2.csv", header = TRUE, sep = ";")
#Some housekeeping bal$Site <- factor(bal$Site) bal$Sample <- factor(bal$Sample) bal$Substrate_ID <- factor(bal$Substrate_ID)
Site -точка сбора материалаSample - квдарат 1х1 м, на котором производился сбор друзBorN - количество Boreotrophon clathratus на квадратеSubstrate_ID - Номер друзыALength - Диаметр апертурыAge - Возраст балянусаPosition - Расположение балянуса (первичный субстрат/вторичный субстрат)Status - живой/мертвый
Drill - Зависимая переменная (0 - нет следов сверления; 1 - есть следы сверления)
Вычислите какая доля живых и мертвых особей несет следы сверления
bal1 <- bal[bal$Status == "live_barnacle", ] mean(bal1$Drill ==1)
## [1] 0.0128
bal2 <- bal[bal$Status == "empty_test", ] mean(bal2$Drill == 1)
## [1] 0.0816
Fix_effect <- formula(Drill ~ BorN + ALength + Age + Position + Site)
Rand_effect <- formula(~1|Sample/Substrate_ID)
Функция максимального правдоподобия для GLMM
\[Lik(\beta, D) = \Pi_i\int f(Y_i|b_i)\times f(b_i)db_i\]
Вычисление максимума функции правдоподобия для GLMM может производиться только в численном виде (аналитическое решение невозможно). Поэтому все алгоритмы очень затратны по времени.
R, позволяющие подобрать GLMM| Пакет | Функция | Особенности работы функции |
|---|---|---|
MASS |
glmPQL() |
Использует penalised quasi-likelihood (PQL) алгоритм, следовательно не выдает AIC. Выбор оптмальной модели может производиться только на основе оценок статистической значимсоти параметров (критерий Вальда). Работает быстро. |
glmmML |
glmmML() |
Выдает значение AIC. Может использовать только один уровень группирующих факторов. Работает быстро. |
lme4 |
glmer() |
Выдает значение AIC. Работает медленно. При сложных моделях часто не сходится. |
glmmADMB |
glmmadmb() |
Выдает значение AIC. Работает ОЧЕНЬ медленно (Для сложных моделей и бльших объемов данных до нескольких часов). |
Важно!Во всех случаях надо с осторожостью принимать решения при уровнях значимости близкх к 5%!
Альтернативный подход к построению сложных моделей - использование методов Байесовской статистики
glmmPQLlibrary(MASS)
M1_PQL <- glmmPQL(Fix_effect, random = ~1|Sample/Substrate_ID,
data = bal2,
family = "binomial")
summary(M1_PQL)
## Linear mixed-effects model fit by maximum likelihood ## Data: bal2 ## AIC BIC logLik ## NA NA NA ## ## Random effects: ## Formula: ~1 | Sample ## (Intercept) ## StdDev: 0.358 ## ## Formula: ~1 | Substrate_ID %in% Sample ## (Intercept) Residual ## StdDev: 1.62 0.704 ## ## Variance function: ## Structure: fixed weights ## Formula: ~invwt ## Fixed effects: Drill ~ BorN + ALength + Age + Position + Site ## Value Std.Error DF t-value p-value ## (Intercept) -4.12 0.445 1908 -9.25 0.0000 ## BorN 0.07 0.053 6 1.35 0.2243 ## ALength 0.15 0.046 1908 3.24 0.0012 ## Age -0.10 0.077 1908 -1.29 0.1969 ## Positionsecondary 1.26 0.172 1908 7.34 0.0000 ## Site2 -0.30 0.612 6 -0.49 0.6437 ## Correlation: ## (Intr) BorN ALngth Age Pstnsc ## BorN -0.776 ## ALength -0.232 -0.035 ## Age 0.079 0.043 -0.907 ## Positionsecondary -0.314 0.047 0.025 0.169 ## Site2 -0.480 0.390 -0.021 0.042 0.069 ## ## Standardized Within-Group Residuals: ## Min Q1 Med Q3 Max ## -4.244 -0.366 -0.205 -0.116 13.809 ## ## Number of Observations: 2120 ## Number of Groups: ## Sample Substrate_ID %in% Sample ## 9 209
glmmML()library(glmmML) M1_ML <- glmmML(Fix_effect, cluster = Substrate_ID, data = bal2) M2_ML <- glmmML(Fix_effect, cluster = Sample, data = bal2) # Коэффициенты C_glmmML_1 <- round(as.numeric(coefficients(M1_ML)), 3) C_glmmML_2 <- round(as.numeric(coefficients(M2_ML)), 3)
glmmML::summary.glmmML(M1_ML)
## ## Call: glmmML(formula = Fix_effect, data = bal2, cluster = Substrate_ID) ## ## ## coef se(coef) z Pr(>|z|) ## (Intercept) -4.3674 0.4100 -10.653 0.000000000 ## BorN 0.0869 0.0405 2.149 0.032000000 ## ALength 0.1316 0.0609 2.162 0.031000000 ## Age -0.0677 0.1036 -0.653 0.510000000 ## Positionsecondary 1.2967 0.2289 5.664 0.000000015 ## Site2 -0.2901 0.5432 -0.534 0.590000000 ## ## Scale parameter in mixing distribution: 1.42 gaussian ## Std. Error: 0.164 ## ## LR p-value for H_0: sigma = 0: 4.77e-23 ## ## Residual deviance: 1020 on 2113 degrees of freedom AIC: 1030
glmmML::summary.glmmML(M2_ML)
## ## Call: glmmML(formula = Fix_effect, data = bal2, cluster = Sample) ## ## ## coef se(coef) z Pr(>|z|) ## (Intercept) -4.1523 0.4256 -9.757 0.0e+00 ## BorN 0.1173 0.0491 2.388 1.7e-02 ## ALength 0.0652 0.0515 1.266 2.1e-01 ## Age 0.0360 0.0859 0.419 6.8e-01 ## Positionsecondary 1.2242 0.1838 6.661 2.7e-11 ## Site2 0.2051 0.5305 0.387 7.0e-01 ## ## Scale parameter in mixing distribution: 0.458 gaussian ## Std. Error: 0.137 ## ## LR p-value for H_0: sigma = 0: 0.00000327 ## ## Residual deviance: 1090 on 2113 degrees of freedom AIC: 1110
glmer()library(lme4)
M1_glmer <- glmer(Drill ~ BorN + ALength + Age + Position + Site +
(1|Sample/Substrate_ID), data = bal2,
family = "binomial")
C_glmer <- round(lme4::fixef(M1_glmer), 3)
summary(M1_glmer)
## Generalized linear mixed model fit by maximum likelihood (Laplace ## Approximation) [glmerMod] ## Family: binomial ( logit ) ## Formula: ## Drill ~ BorN + ALength + Age + Position + Site + (1 | Sample/Substrate_ID) ## Data: bal2 ## ## AIC BIC logLik deviance df.resid ## 1033 1078 -508 1017 2112 ## ## Scaled residuals: ## Min 1Q Median 3Q Max ## -2.399 -0.262 -0.163 -0.101 9.177 ## ## Random effects: ## Groups Name Variance Std.Dev. ## Substrate_ID:Sample (Intercept) 1.8578 1.363 ## Sample (Intercept) 0.0634 0.252 ## Number of obs: 2120, groups: Substrate_ID:Sample, 209; Sample, 9 ## ## Fixed effects: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -4.4040 0.4510 -9.76 < 2e-16 *** ## BorN 0.0941 0.0479 1.96 0.050 * ## ALength 0.1270 0.0614 2.07 0.039 * ## Age -0.0605 0.1042 -0.58 0.561 ## Positionsecondary 1.3065 0.2290 5.71 0.000000012 *** ## Site2 -0.2308 0.5862 -0.39 0.694 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## Correlation of Fixed Effects: ## (Intr) BorN ALngth Age Pstnsc ## BorN -0.698 ## ALength -0.295 -0.077 ## Age 0.103 0.086 -0.912 ## Postnscndry -0.433 0.082 0.032 0.148 ## Site2 -0.417 0.396 -0.050 0.074 0.090 ## convergence code: 0 ## Model failed to converge with max|grad| = 0.00113631 (tol = 0.001, component 1)
Parameter glmmPQL glmmML_1 glmmML_2 glmer
1 (Intercept) -4.119 -4.367 -4.152 -4.404
2 BorN 0.072 0.087* 0.117* 0.094*
3 ALength 0.148* 0.132* 0.065 0.127*
4 Age -0.100 -0.068 0.036 -0.061
5 Positionsecondary 1.261* 1.297* 1.224* 1.306*
6 Site2 -0.298 -0.290 0.205 -0.231
drop1(M1_glmer)
## Single term deletions ## ## Model: ## Drill ~ BorN + ALength + Age + Position + Site + (1 | Sample/Substrate_ID) ## Df AIC ## <none> 1033 ## BorN 1 1035 ## ALength 1 1035 ## Age 1 1031 ## Position 1 1064 ## Site 1 1031
M2_glmer <- update(M1_glmer, .~.- Site) anova(M1_glmer, M2_glmer)
## Data: bal2 ## Models: ## M2_glmer: Drill ~ BorN + ALength + Age + Position + (1 | Sample/Substrate_ID) ## M1_glmer: Drill ~ BorN + ALength + Age + Position + Site + (1 | Sample/Substrate_ID) ## Df AIC BIC logLik deviance Chisq Chi Df Pr(>Chisq) ## M2_glmer 7 1031 1071 -509 1017 ## M1_glmer 8 1033 1078 -508 1017 0.15 1 0.69
drop1(M2_glmer)
## Single term deletions ## ## Model: ## Drill ~ BorN + ALength + Age + Position + (1 | Sample/Substrate_ID) ## Df AIC ## <none> 1031 ## BorN 1 1034 ## ALength 1 1033 ## Age 1 1029 ## Position 1 1063
M3_glmer <- update(M2_glmer, .~.-Age) anova(M2_glmer, M3_glmer)
## Data: bal2 ## Models: ## M3_glmer: Drill ~ BorN + ALength + Position + (1 | Sample/Substrate_ID) ## M2_glmer: Drill ~ BorN + ALength + Age + Position + (1 | Sample/Substrate_ID) ## Df AIC BIC logLik deviance Chisq Chi Df Pr(>Chisq) ## M3_glmer 6 1029 1063 -509 1017 ## M2_glmer 7 1031 1071 -509 1017 0.31 1 0.58
drop1(M3_glmer)
## Single term deletions ## ## Model: ## Drill ~ BorN + ALength + Position + (1 | Sample/Substrate_ID) ## Df AIC ## <none> 1029 ## BorN 1 1032 ## ALength 1 1042 ## Position 1 1064
summary(M3_glmer)
## Generalized linear mixed model fit by maximum likelihood (Laplace ## Approximation) [glmerMod] ## Family: binomial ( logit ) ## Formula: ## Drill ~ BorN + ALength + Position + (1 | Sample/Substrate_ID) ## Data: bal2 ## ## AIC BIC logLik deviance df.resid ## 1029 1063 -509 1017 2114 ## ## Scaled residuals: ## Min 1Q Median 3Q Max ## -2.371 -0.262 -0.163 -0.103 9.193 ## ## Random effects: ## Groups Name Variance Std.Dev. ## Substrate_ID:Sample (Intercept) 1.8092 1.345 ## Sample (Intercept) 0.0773 0.278 ## Number of obs: 2120, groups: Substrate_ID:Sample, 209; Sample, 9 ## ## Fixed effects: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -4.4498 0.4108 -10.83 < 2e-16 *** ## BorN 0.1035 0.0450 2.30 0.02154 * ## ALength 0.0948 0.0252 3.76 0.00017 *** ## Positionsecondary 1.3335 0.2256 5.91 0.0000000034 *** ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## Correlation of Fixed Effects: ## (Intr) BorN ALngth ## BorN -0.663 ## ALength -0.517 -0.015 ## Postnscndry -0.462 0.040 0.411
plot(M3_glmer)