class: top, center, inverse, title-slide # Кластерный анализ ## Анализ и визуализация многомерных данных с использованием R ### Марина Варфоломеева ### Вадим Хайтов --- ## Кластерный анализ - Методы построения деревьев - Методы кластеризации на основании расстояний - Примеры для демонстрации и для заданий - Кластерный анализ в R - Качество кластеризации: - кофенетическая корреляция - ширина силуэта - поддержка ветвей - Сопоставление деревьев: танглграммы ### Вы сможете - Выбирать подходящий метод агрегации (алгоритм кластеризации) - Строить дендрограммы - Оценивать качество кластеризации (Кофенетическая корреляция, ширина силуэта, поддержка ветвей) - Сопоставлять дендрограммы, полученные разными способами, при помощи танглграмм --- class: middle, center, inverse # Кластерный анализ --- ## Какие бывают методы построения деревьев? ### Методы кластеризации на основании расстояний (о них сегодня) - Метод ближайшего соседа - Метод отдаленного соседа - Метод среднегруппового расстояния - Метод Варда - и т.д. и т.п. ### Методы кластеризации на основании признаков - Метод максимальной бережливости - Метод максимального правдоподобия ### И это еще далеко не все --- class: middle, center, inverse # Пример: Волки --- ## Пример: Волки Морфометрия черепов у волков в Скалистых горах и в Арктике (Jolicoeur, 1959)  Данные взяты из работы Morrison (1990): - A --- волки из Арктики (10 самцов, 6 самок) - L --- волки из Скалистых гор (6 самцов, 3 самки) ```r library(candisc) data("Wolves") ``` --- ## Знакомимся с данными ```r dim(Wolves) ``` ``` ## [1] 25 12 ``` ```r colnames(Wolves) ``` ``` ## [1] "group" "location" "sex" "x1" "x2" "x3" ## [7] "x4" "x5" "x6" "x7" "x8" "x9" ``` ```r head(rownames(Wolves)) ``` ``` ## [1] "rmm1" "rmm2" "rmm3" "rmm4" "rmm5" "rmm6" ``` ```r any(is.na(Wolves)) ``` ``` ## [1] FALSE ``` ```r table(Wolves$group) ``` ``` ## ## ar:f ar:m rm:f rm:m ## 6 10 3 6 ``` --- ## Задание - Постройте ординацию nMDS данных - Оцените качество ординации - Обоснуйте выбор коэффициента - Раскрасьте точки на ординации волков в зависимости от географического происхождения (`group`) --- ## Решение .scroll-box-20[ ```r library(vegan) library(ggplot2); theme_set(theme_bw(base_size = 16)) s_w <- scale(Wolves[, 4:ncol(Wolves)]) ## стандартизируем ord_w <- metaMDS(comm = s_w, distance = "euclidean", autotransform = FALSE) dfr_w <- data.frame(ord_w$points, Group = Wolves$group) gg_w <- ggplot(dfr_w, aes(x = MDS1, y = MDS2)) + geom_point(aes(colour = Group)) ``` ``` ## Run 0 stress 0.101 ## Run 1 stress 0.128 ## Run 2 stress 0.128 ## Run 3 stress 0.126 ## Run 4 stress 0.136 ## Run 5 stress 0.101 ## ... New best solution ## ... Procrustes: rmse 0.00000401 max resid 0.0000114 ## ... Similar to previous best ## Run 6 stress 0.127 ## Run 7 stress 0.156 ## Run 8 stress 0.101 ## ... Procrustes: rmse 0.00000411 max resid 0.0000136 ## ... Similar to previous best ## Run 9 stress 0.137 ## Run 10 stress 0.101 ## ... Procrustes: rmse 0.00000633 max resid 0.0000208 ## ... Similar to previous best ## Run 11 stress 0.127 ## Run 12 stress 0.127 ## Run 13 stress 0.101 ## ... Procrustes: rmse 0.00000696 max resid 0.000023 ## ... Similar to previous best ## Run 14 stress 0.137 ## Run 15 stress 0.128 ## Run 16 stress 0.101 ## ... New best solution ## ... Procrustes: rmse 0.00000302 max resid 0.00000761 ## ... Similar to previous best ## Run 17 stress 0.101 ## ... Procrustes: rmse 0.00000467 max resid 0.0000131 ## ... Similar to previous best ## Run 18 stress 0.127 ## Run 19 stress 0.136 ## Run 20 stress 0.147 ## *** Solution reached ``` ] --- ## Решение <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-4-1.png" style="display: block; margin: auto;" /> --- class: middle, center, inverse # Методы кластеризации на основании расстояний --- ## Этапы кластеризации .pull-left[ <div id="htmlwidget-f62c96e0ee521dc7a21c" style="width:432px;height:504px;" class="DiagrammeR html-widget"></div> <script type="application/json" data-for="htmlwidget-f62c96e0ee521dc7a21c">{"x":{"diagram":"graph TD; \n A(Набор признаков)-->B(Матрица расстояний или сходств); \n B-->C(Группировка объектов);\n C-->D(Отношения между кластерами); D-->E(Поправки); E-->A;\n "},"evals":[],"jsHooks":[]}</script> ] .pull-right[ Результат кластеризации зависит от - выбора признаков - коэффициента сходства-различия - от алгоритма кластеризации ] --- ## Методы кластеризации <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-6-1.png" style="display: block; margin: auto;" /> --- ## Метод ближайшего соседа .pull-left-66[ .content-box-purple[ ### = nearest neighbour = single linkage - к кластеру присоединяется ближайший к нему кластер/объект - кластеры объединяются в один на расстоянии, которое равно расстоянию между ближайшими объектами этих кластеров ] ] .pull-right-33[ <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-7-1.png" style="display: block; margin: auto;" /> ] ### Особенности - Может быть сложно интерпретировать, если нужны группы - объекты на дендрограмме часто не образуют четко разделенных групп - часто получаются цепочки кластеров (объекты присоединяются как бы по-одному) - Хорош для выявления градиентов --- ## Как работает метод ближайшего соседа <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-8-1.png" style="display: block; margin: auto;" /> --- ## Как работает метод ближайшего соседа <img src="05_cluster_analysis_files/figure-html/single-ani-.gif" style="display: block; margin: auto;" /> --- ## Метод отдаленного соседа .pull-left-66[ .content-box-purple[ ### = furthest neighbour = complete linkage - к кластеру присоединяется отдаленный кластер/объект - кластеры объединяются в один на расстоянии, которое равно расстоянию между самыми отдаленными объектами этих кластеров (следствие - чем более крупная группа, тем сложнее к ней присоединиться) ]] .pull-right-33[ <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-9-1.png" style="display: block; margin: auto;" /> ] ### Особенности - На дендрограмме образуется много отдельных некрупных групп - Хорош для поиска дискретных групп в данных --- ## Как работает метод отдаленного соседа <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-10-1.png" style="display: block; margin: auto;" /> --- ## Как работает метод отдаленного соседа <img src="05_cluster_analysis_files/figure-html/complete-ani-.gif" style="display: block; margin: auto;" /> --- ## Метод невзвешенного попарного среднего .pull-left-66[ .content-box-purple[ ### = UPGMA = Unweighted Pair Group Method with Arithmetic mean - кластеры объединяются в один на расстоянии, которое равно среднему значению всех возможных расстояний между объектами из разных кластеров. ]] .pull-right-33[ <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-11-1.png" style="display: block; margin: auto;" /> ] ### Особенности - UPGMA и WUPGMС иногда могут приводить к инверсиям на дендрограммах .pull-left-66[ 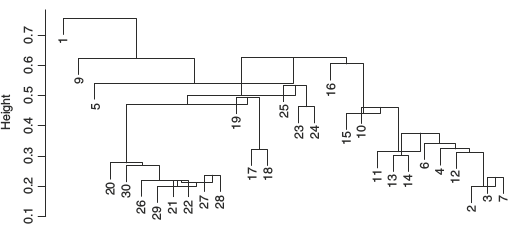 ] .pull-right-33[ .pull-down[ из Borcard et al., 2011 ]] --- ## Как работает метод среднегруппового расстояния <img src="05_cluster_analysis_files/figure-html/average-ani-.gif" style="display: block; margin: auto;" /> --- ## Метод Варда .pull-left-66[ .content-box-purple[ ### = Ward's Minimum Variance Clustering - объекты объединяются в кластеры так, чтобы внутригрупповая дисперсия расстояний была минимальной ]] .pull-right-33[ <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-12-1.png" style="display: block; margin: auto;" /> ] ### Особенности - метод годится и для неевклидовых расстояний несмотря на то, что внутригрупповая дисперсия расстояний рассчитывается так, как будто это евклидовы расстояния --- ## Как работает метод Варда <img src="05_cluster_analysis_files/figure-html/ward-ani-.gif" style="display: block; margin: auto;" /> --- class: middle, center, inverse # Кластерный анализ в R --- ## Кластеризация Давайте построим деревья при помощи нескольких алгоритмов кластеризации (по стандартизованным данным, с использованием Евклидова расстояния) и сравним их. ```r # Матрица расстояний d <- dist(x = s_w, method = "euclidean") # Пакеты для визуализации кластеризации library(ape) library(dendextend) ``` --- ## (1.0) Метод ближайшего соседа + `base` ```r hc_single <- hclust(d, method = "single") plot(hc_single) ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-14-1.png" style="display: block; margin: auto;" /> --- ## (1.1) Метод ближайшего соседа + `ape` ```r ph_single <- as.phylo(hc_single) plot(ph_single, type = "phylogram") axisPhylo() ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-15-1.png" style="display: block; margin: auto;" /> --- ## (1.2) Метод ближайшего соседа + `dendextend` ```r den_single <- as.dendrogram(hc_single) plot(den_single) ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-16-1.png" style="display: block; margin: auto;" /> --- ## (2.1) Метод отдаленного соседа + `ape` ```r hc_compl <- hclust(d, method = "complete") ph_compl <- as.phylo(hc_compl) plot(ph_compl, type = "phylogram") axisPhylo() ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-17-1.png" style="display: block; margin: auto;" /> --- ## (2.2)Метод отдаленного соседа + `dendextend` ```r den_compl <- as.dendrogram(hc_compl) plot(den_compl) ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-18-1.png" style="display: block; margin: auto;" /> --- ## (3.1) Метод невзвешенного попарного среднего + `ape` ```r hc_avg <- hclust(d, method = "average") ph_avg <- as.phylo(hc_avg) plot(ph_avg, type = "phylogram") axisPhylo() ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-19-1.png" style="display: block; margin: auto;" /> --- ## (3.2) Метод невзвешенного попарного среднего + `dendextend` ```r den_avg <- as.dendrogram(hc_avg) plot(den_avg) ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-20-1.png" style="display: block; margin: auto;" /> --- ## (4.1) Метод Варда + `ape` ```r hc_w2 <-hclust(d, method = "ward.D2") ph_w2 <- as.phylo(hc_w2) plot(ph_w2, type = "phylogram") axisPhylo() ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-21-1.png" style="display: block; margin: auto;" /> --- ## (4.2) Метод Варда + `dendextend` ```r den_w2 <- as.dendrogram(hc_w2) plot(den_w2) ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-22-1.png" style="display: block; margin: auto;" /> --- class: middle, center, inverse # Качество кластеризации --- ## Кофенетическая корреляция __Кофенетическое расстояние__ - расстояние между объектами на дендрограмме .content-box-purple[ __Кофенетическая корреляция__ - мера качества отображения многомерных данных на дендрограмме. Кофенетическую корреляцию можно рассчитать как Пирсоновскую корреляцию (обычную) между матрицами исходных и кофенетических расстояний между всеми парами объектов ] <br/> Метод агрегации, который дает наибольшую кофенетическую корреляцию, дает кластеры, лучше всего отражающие исходные данные Можно рассчитать при помощи функции из пакета `ape` --- ## Кофенетическая корреляция в R ```r # Матрица кофенетических расстояний c_single <- cophenetic(ph_single) # Кофенетическая корреляция = # = корреляция матриц кофенетич. и реальн. расстояний cor(d, as.dist(c_single)) ``` ``` ## [1] 0.565 ``` --- ## Задание: Оцените при помощи кофенетической корреляции качество кластеризаций, полученных разными методами. Какой метод дает лучший результат? --- ## Решение: ```r c_single <- cophenetic(ph_single) cor(d, as.dist(c_single)) ``` ``` ## [1] 0.565 ``` ```r c_compl <- cophenetic(ph_compl) cor(d, as.dist(c_compl)) ``` ``` ## [1] 0.706 ``` ```r c_avg <- cophenetic(ph_avg) cor(d, as.dist(c_avg)) ``` ``` ## [1] 0.745 ``` ```r c_w2 <- cophenetic(ph_w2) cor(d, as.dist(c_w2)) ``` ``` ## [1] 0.726 ``` --- ## Что можно делать дальше с дендрограммой - Можно выбрать число кластеров: + либо субъективно, на любом выбранном уровне (главное, чтобы кластеры были осмысленными и интерпретируемыми); + либо исходя из распределения расстояний ветвления. - Можно оценить стабильность кластеризации при помощи бутстрепа. --- ## Ширина силуэта .content-box-purple[ Ширина силуэта `\(s_i\)` --- мера степени принадлежности объекта `\(i\)` к кластеру ] `$$s_i = \frac {\color{purple}{\bar{d}_{i~to~nearest~cluster}} - \color{green}{\bar{d}_{i~within}}} {max\{\color{purple}{\bar{d}_{i~to~nearest~cluster}}, \color{green}{\bar{d}_{i~within}}\}}$$` `\(s_i\)` --- сравнивает между собой средние расстояния от данного объекта: - `\(\color{green} {\bar{d}_{i~within}}\)` --- до других объектов из того же кластера - `\(\color{purple} {\bar{d}_{i~to~nearest~cluster}}\)` --- до ближайшего кластера `\(-1 \le s_i \le 1\)` --- чем больше `\(s_i\)`, тем "лучше" объект принадлежит кластеру. - Средняя ширина силуэта для всех объектов из кластера --- оценивает, насколько "тесно" сгруппированы объекты. - Средняя ширина силуэта по всем данным --- оценивает общее качество классификации. - Чем больше к 1, тем лучше. Если меньше 0.25, то можно сказать, что нет структуры. --- ## Как рассчитывается ширина силуэта Оценим ширину силуэта для 3 кластеров ```r library(cluster) complete3 <- cutree(tree = hc_avg, k = 3) plot(silhouette(x = complete3, dist = d), cex.names = 0.6) ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-25-1.png" style="display: block; margin: auto;" /> --- ## Бутстреп поддержка ветвей "An approximately unbiased test of phylogenetic tree selection" (Shimodaria, 2002) Этот тест использует специальный вариант бутстрепа --- multiscale bootstrap. Мы не просто многократно берем бутстреп-выборки и оцениваем для них вероятность получения топологий (BP p-value), эти выборки еще и будут с разным числом объектов. По изменению BP при разных объемах выборки можно вычислить AU (approximately unbiased p-value). ```r library(pvclust) # итераций должно быть 1000 и больше, здесь мало для скорости cl_boot <- pvclust(t(s_w), method.hclust = "average", nboot = 100, method.dist = "euclidean", parallel = TRUE, iseed = 42) ``` ``` ## Creating a temporary cluster...done: ## socket cluster with 7 nodes on host 'localhost' ## Multiscale bootstrap... Done. ``` Обратите внимание на число итераций: `nboot = 100` --- это очень мало. На самом деле нужно 10000 или больше. --- ## Дерево с величинами поддержки AU --- approximately unbiased p-values (красный), BP --- bootstrap p-values (зеленый) ```r plot(cl_boot) # pvrect(cl_boot) # достоверные ветвления ``` <img src="05_cluster_analysis_files/figure-html/fig-cl-boot-1.png" style="display: block; margin: auto;" /> --- ## Для диагностики качества оценок AU График стандартных ошибок для AU p-value нужен, чтобы оценить точность оценки самих AU. Чем больше было бутстреп-итераций, тем точнее будет оценка AU. ```r seplot(cl_boot) # print(cl_boot) # все значения ``` <img src="05_cluster_analysis_files/figure-html/fig-seplot-1.png" style="display: block; margin: auto;" /> --- class: middle, center, inverse # Сопоставление деревьев: Танглграммы --- ## Танглграмма Два дерева (с непохожим ветвлением) выравнивают, вращая случайным образом ветви вокруг оснований. Итеративный алгоритм. Картина каждый раз разная. ```r set.seed(395) untang_w <- untangle_step_rotate_2side(den_compl, den_w2, print_times = F) # танглграмма tanglegram(untang_w[[1]], untang_w[[2]], highlight_distinct_edges = FALSE, common_subtrees_color_lines = F, main = "Tanglegram", main_left = "Left tree", main_right = "Right tree", columns_width = c(8, 1, 8), margin_top = 3.2, margin_bottom = 2.5, margin_inner = 4, margin_outer = 0.5, lwd = 1.2, edge.lwd = 1.2, lab.cex = 1.5, cex_main = 2) ``` --- ## Танглграмма <img src="05_cluster_analysis_files/figure-html/tang-1.png" style="display: block; margin: auto;" /> --- ## Задание Постройте танглграмму из дендрограмм, полученных методом ближайшего соседа и методом Варда. --- class: middle, center, inverse # Деревья по генетическим данным --- ## И небольшая демонстрация - дерево по генетическим данным ```r webpage <-"http://evolution.genetics.washington.edu/book/primates.dna" primates.dna <- read.dna(webpage) d_pri <- dist.dna(primates.dna) hc_pri <- hclust(d_pri, method = "average") ph_pri <- as.phylo(hc_pri) plot(ph_pri) axisPhylo() ``` <img src="05_cluster_analysis_files/figure-html/unnamed-chunk-27-1.png" style="display: block; margin: auto;" /> --- ## Take-home messages - Результат кластеризации зависит не только от выбора коэффициента, но и от выбора алгоритма. - Качество кластеризации можно оценить разными способами. - Кластеризации, полученные разными методами, можно сравнить на танглграммах. --- ## Дополнительные ресурсы - Borcard, D., Gillet, F., Legendre, P., 2011. Numerical ecology with R. Springer. - Legendre, P., Legendre, L., 2012. Numerical ecology. Elsevier. - Quinn, G.G.P., Keough, M.J., 2002. Experimental design and data analysis for biologists. Cambridge University Press. --- ## И еще ресурсы Как работает UPGMA можно посмотреть здесь: - http://www.southampton.ac.uk/~re1u06/teaching/upgma/ Как считать поддержку ветвей (пакет + статья): - pvclust: An R package for hierarchical clustering with p-values [WWW Document], n.d. URL http://www.sigmath.es.osaka-u.ac.jp/shimo-lab/prog/pvclust/ (accessed 11.7.14). Для анализа молекулярных данных: - Paradis, E., 2011. Analysis of Phylogenetics and Evolution with R. Springer. --- ## Данные для самостоятельной работы ### Фораминиферы маршей Белого моря (Golikova et al. 2020) 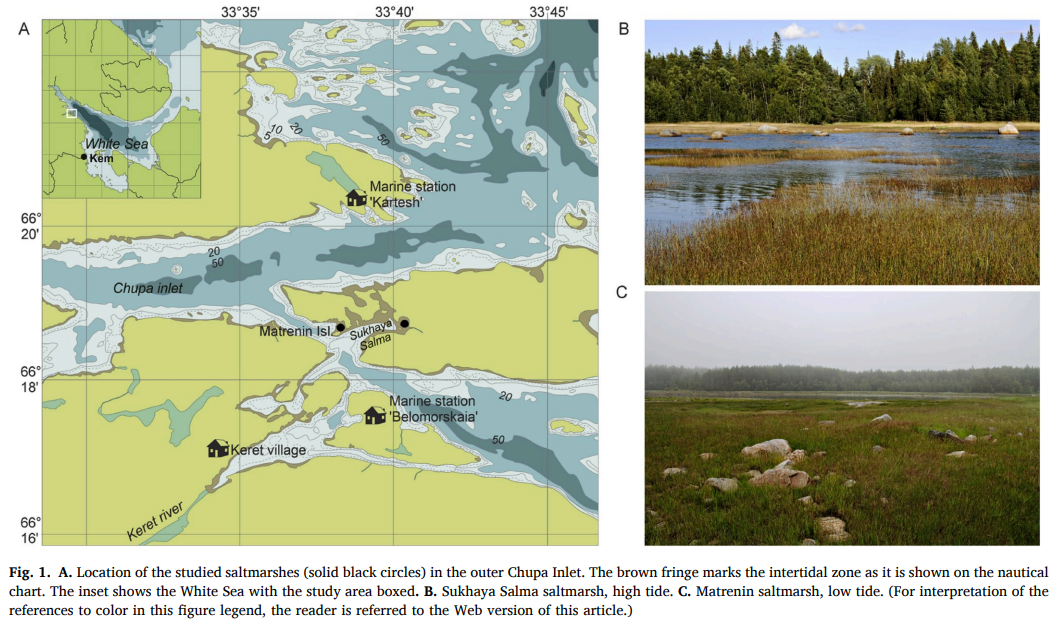 --- ## Фораминиферы маршей Белого моря  --- ## Фораминиферы маршей Белого моря .pull-left[  ] .pull-right[ Plate 1. 1. _Balticammina pseudomacrescens_. 2. _Ammotium salsum_. 3. _Jadammina macrescens_. 4. _Trochammina inflata_. 5. _Ammobaculites balkwilli?_ 6. _Miliammina fusca_. 7. _Ovammina opaca_. 8. _Elphidium albiumbilicatum_. 9. _Elphidium williamsoni_. Scale bar 500 μm. ] --- ## Задание Проанализируйте данные об относительных обилиях фораминифер в пробах на Белом море. - Выберите и обоснуйте трансформацию данных и расстояние. - Постройте ординацию nMDS по относительным обилиям фораминифер: - цвет значков --- растение-доминант, - форма значков --- точка сбора. - Постройте дендрограмму проб по сходству отн. обилий фораминифер. - оцените при помощи кофенетической корреляции, какой метод аггрегации лучше, - Опишите получившиеся кластеры при помощи различных параметров: - ширина силуэта - бутстреп-поддержка ветвлений --- ## Фораминиферы маршей Белого моря  --- ## Фораминиферы маршей Белого моря