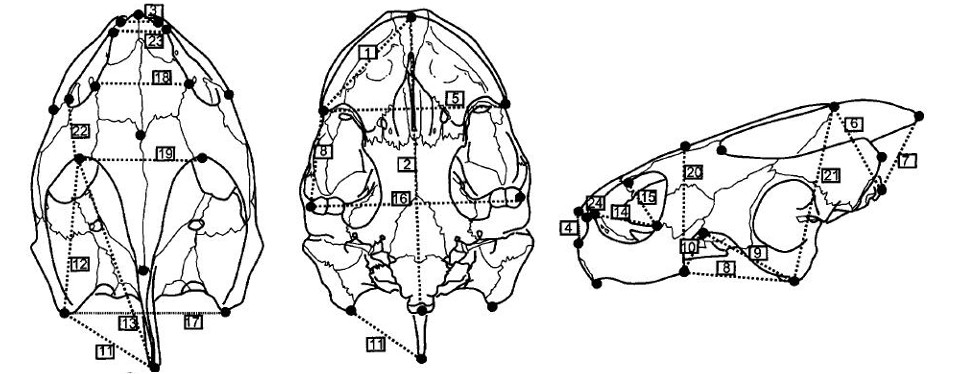
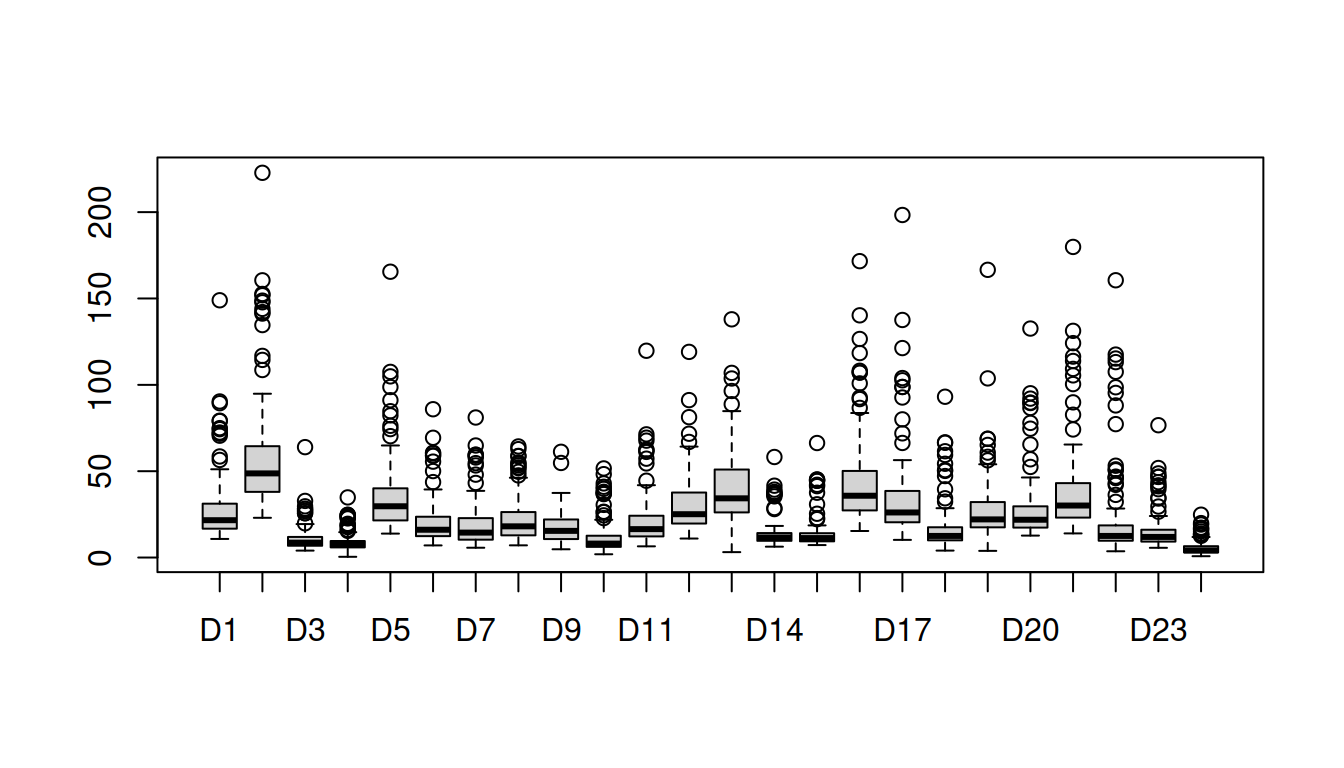
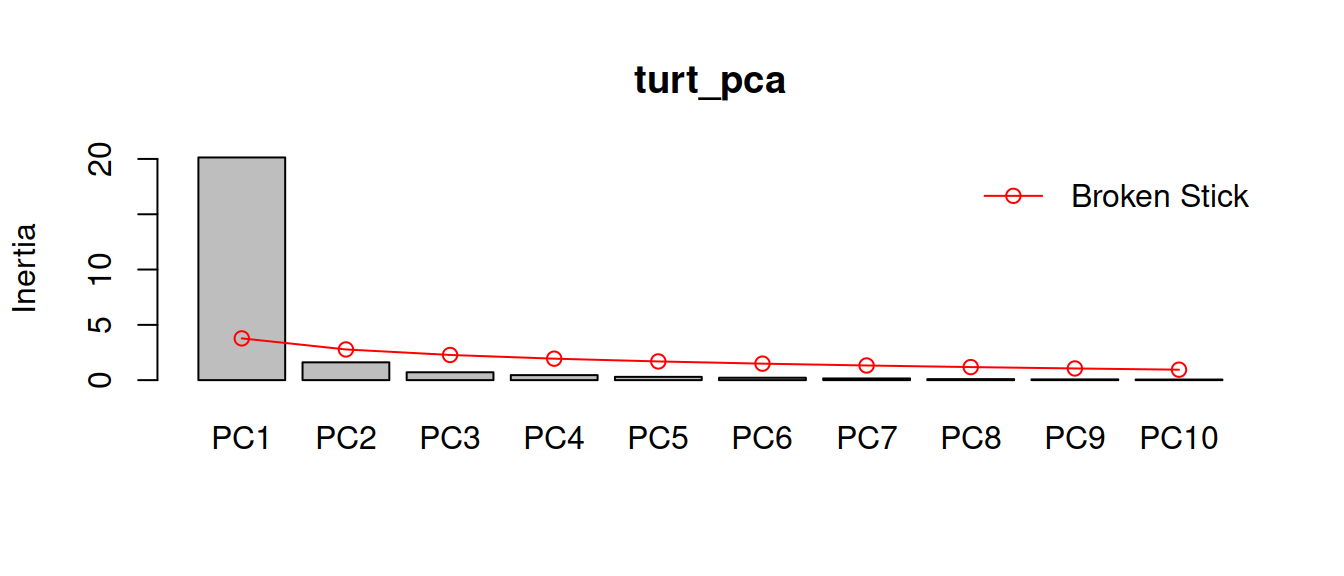
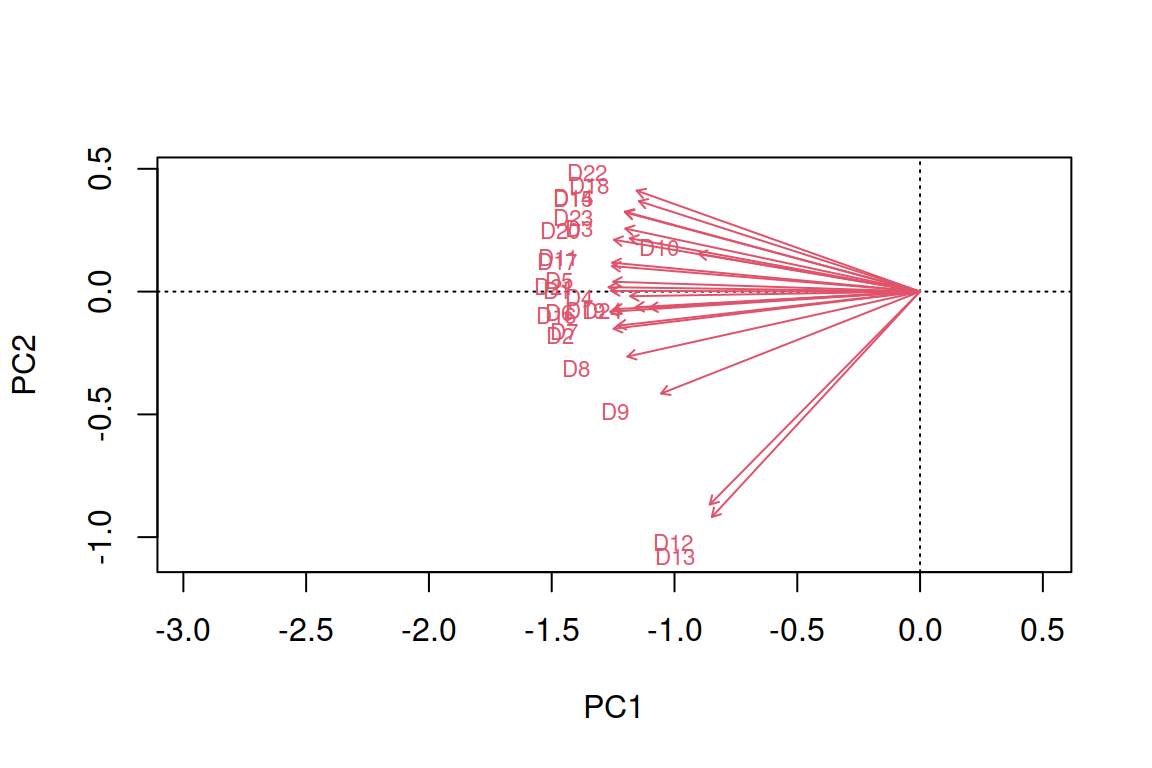
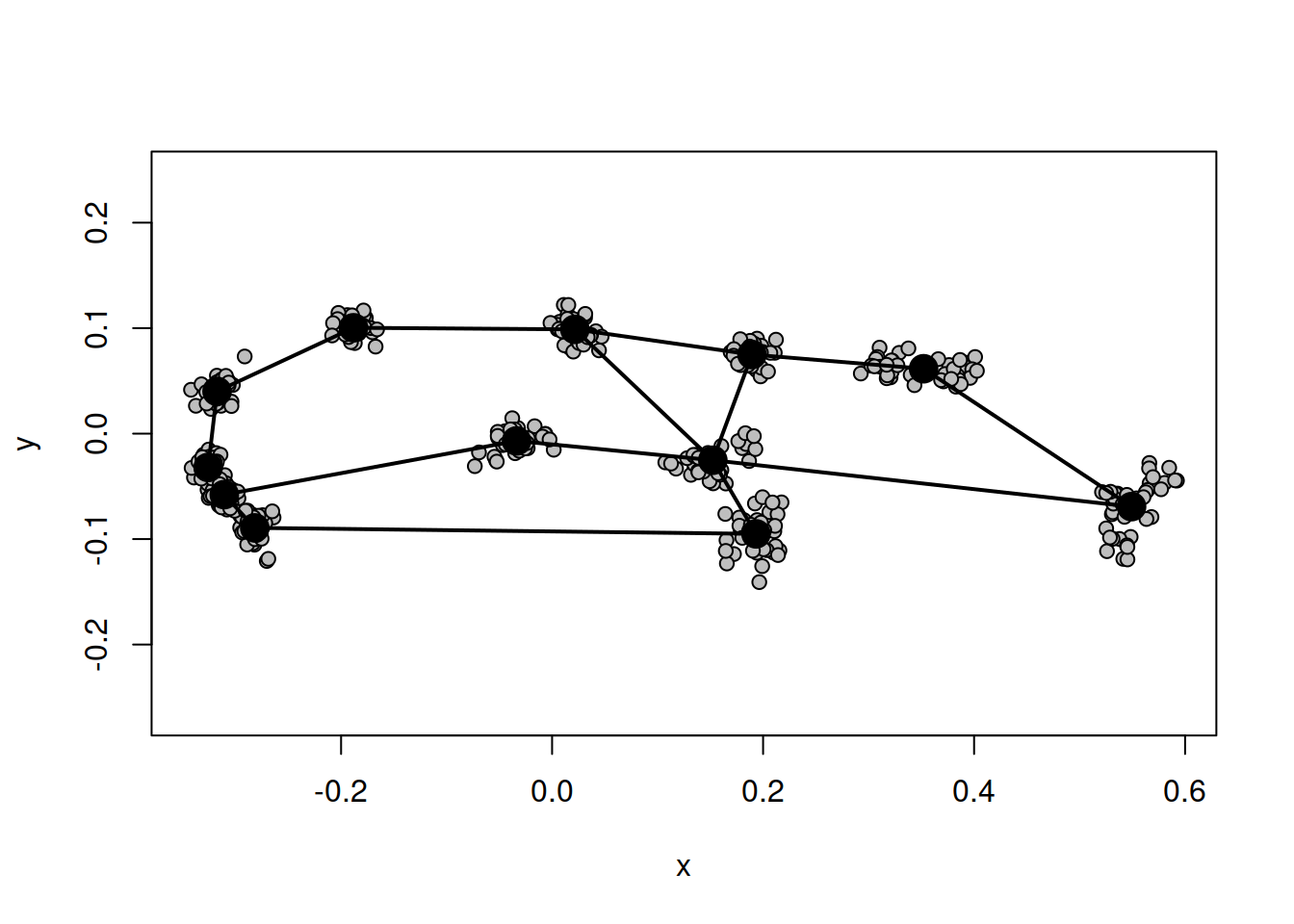
# матрица, в которой хранится разметка общего графика
m <- matrix(data = c(1, 2,
3, 3),
nrow = 2, ncol = 2, byrow = TRUE)
l <- layout(m, heights = c(1, 1), widths = c(1, 1))
# layout.show(l) # можно просмотреть разметку
# Графики
op <- par( mar = c(0, 0, 0, 0))
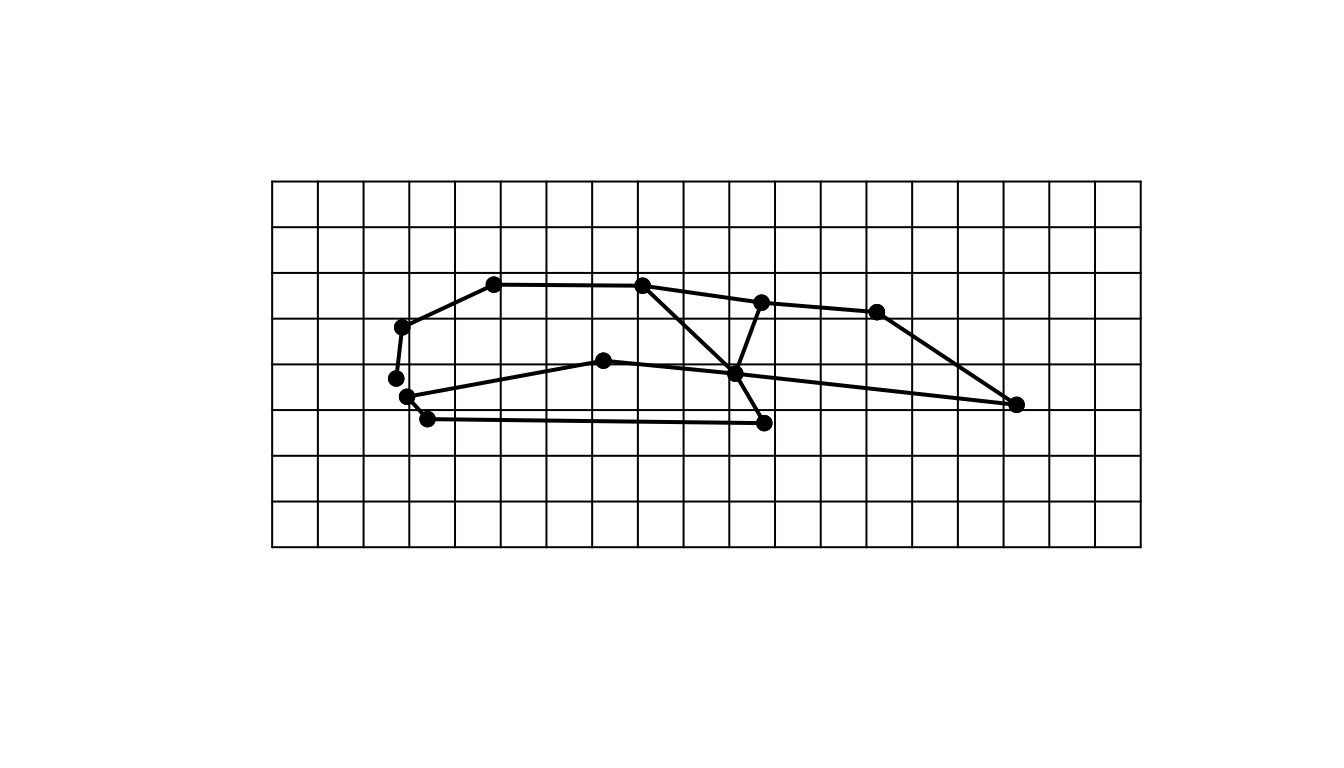
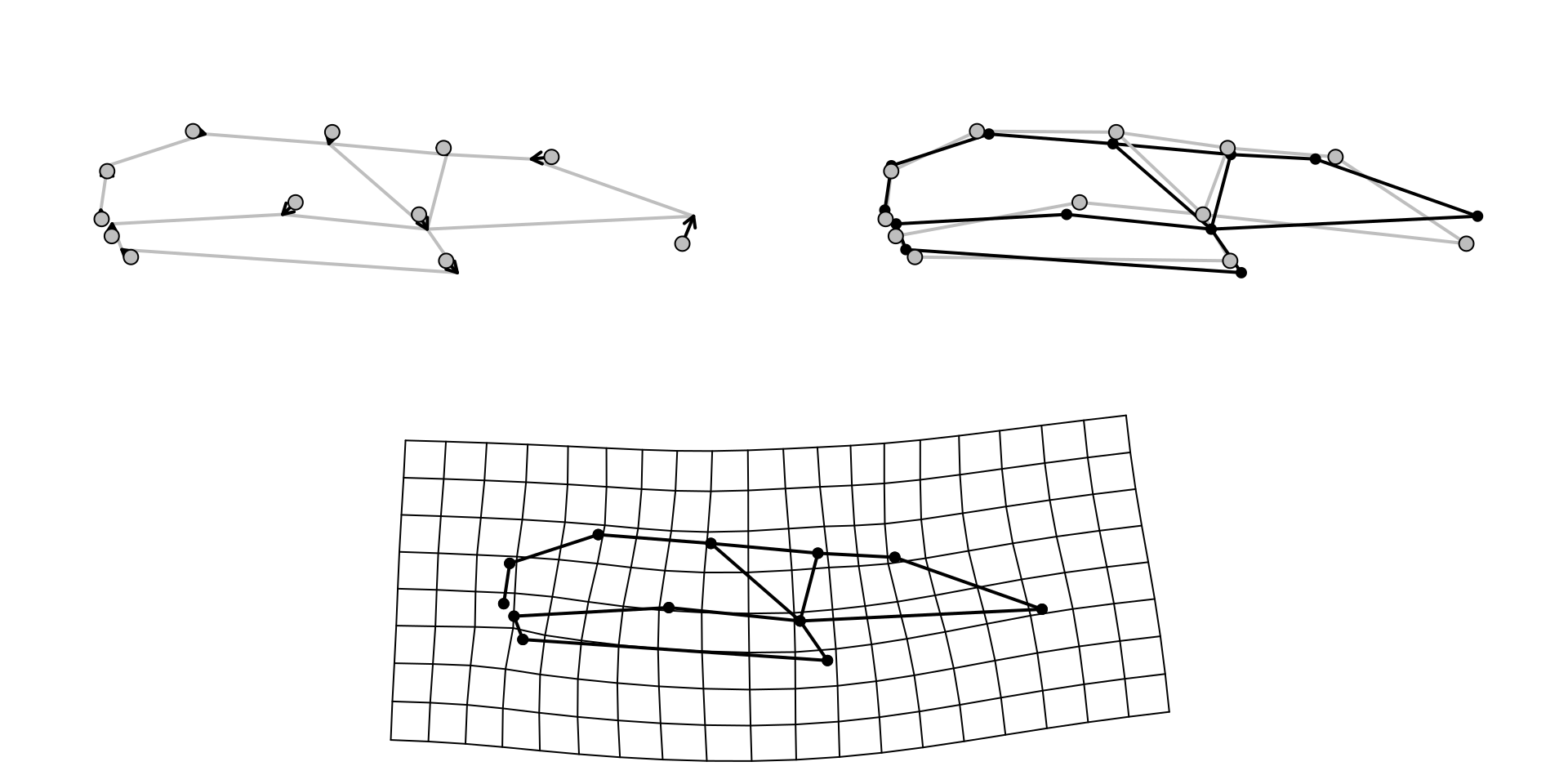
# 1) изменение конфигурации обозначено векторами
plotRefToTarget(ref, gpa$coords[, , 11],
method = "vector", mag = 1,
links = plethodon$links)
# 2) формы обозначены точками
plotRefToTarget(ref, gpa$coords[, , 11],
method = "points", mag = 1,
links = plethodon$links)
# 3) сплайн
plotRefToTarget(ref, gpa$coords[, , 11],
method = "TPS", mag = 1,
links = plethodon$links)
par(op)