О модуле Анализ протеомных данных
Марина Варфоломеева
Дифференциальный двумерный электрофорез [two-dimensional difference gel electrophoresis, 2D-DIGE; Unlü et al. (1997)] доступный и чувствительный метод, который позволяет сравнивать уровень экспрессии белков в различных выборках. Этот метод часто используется в протеомике для выявления биомаркеров.
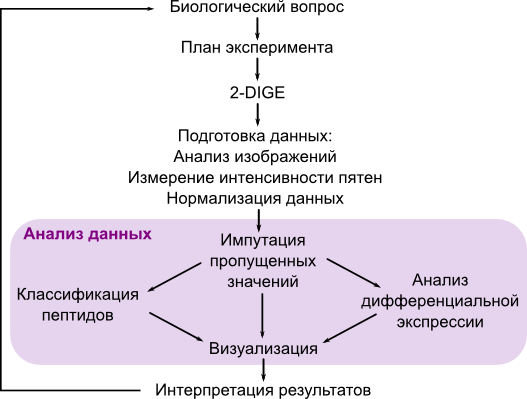
Схема протеомного эксперимента
В этой части курса вы познакомитесь с различными способами анализа данных, полученных протеомными методами. Мы рассмотрим анализ 2-DE (two-dimensional gel electrophoresis) данных с момента, когда информация об интенсивности пятен уже получена (и нормализована), т.е. собственно анализ данных для ответа на биологический вопрос исследования (Sellers and Miecznikowski 2012, Valledor et al. 2014).
Раздел 1 посвящен языку статистического программирования R (R Core Team 2021). Читатели познакомятся с работой в среде RStudio (RStudio Team 2019), освоят основные элементы языка R.
В разделе 2 обсуждаются техники подготовки и предварительной обработки данных протеомных исследований. Мы поговорим о том, в каком формате удобно хранить данные, каким образом их можно использовать в R, мы обсудим проблему пропусков в данных и научимся их заполнять.
Раздел 3 посвящен методам классификации. В нем рассказывается о различных типах расстояний, и алгоритмах, которые используются для кластеризации белков или проб. Читатели научатся строить дендрограммы, оценивать надежность их ветвей, рисовать тепловые карты экспрессии белков.
В разделе 4 говорится о методах сравнения уровня экспрессии в разных пробах или группах проб. Читатель научится выявлять дифференциально экспрессируемые белки при помощи fold change анализа и модерируемого t-критерия. Кроме того, мы обсудим проблемы, возникающие при множественном тестировании и статистические поправки, которые позволяют их избегать.
Раздел 5 рассказывает о выявлении групп маркеров с похожим уровнем экспрессии при помощи анализа главных компонент (Principal Component Analysis, PCA).
Раздел 6 посвящен многомерному анализу паттернов дифференциальной экспрессии при помощи сочетания регрессии методом частных наименьших квадратов и дискриминантного анализа (Partial Least Squares Regression and Discriminant Analysis, PLS-DA).
Заметки к лекциям этого модуля написаны при помощи RMarkdown (Xie et al. 2018, 2020, Allaire et al. 2021) и knitr (Xie 2014, 2015, 2021).
Ссылки
(C) 2019-present Marina Varfolomeeva, Arina Maltseva